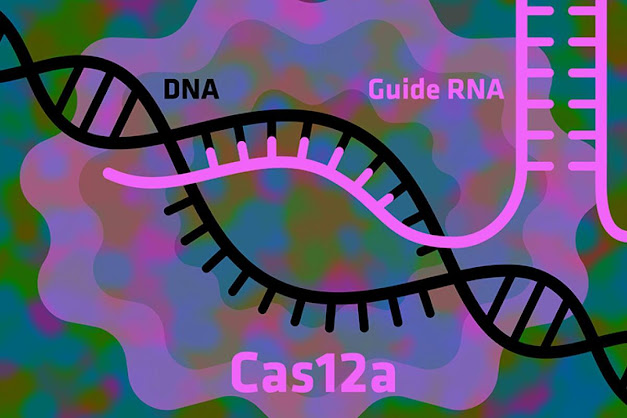
CRISPR/Cas12a
Cas12a (önceden Cpf1 olarak bilinen CRISPR ile ilişkili protein 12a), bazı bakterilerde CRISPR sisteminin bir parçasını oluşturan ve bilim adamları tarafından DNA'yı değiştirmek için kullanılan RNA güdümlü bir endonükleazdır.[1][2] Bakteriyel bağışıklık mekanizmasının bir parçası olarak ortaya çıkar ve burada virüslerin genetik materyalini yok etmeye ve böylece hücreyi ve koloniyi viral enfeksiyondan korumaya hizmet eder. Cas12a ve diğer CRISPR ile ilişkili endonükleazlar, kılavuz RNA kullanımlarının bu eylemi oldukça seçici hale getirmesi bakımından benzersizdir; DNA'yı yalnızca belirli bir nükleotid dizisine bitişik olduğunda keserler. Kaynaklandığı organizmalarda, bu kılavuz RNA, daha önce hücreyi enfekte etmiş bir virüsün genomunun bir parçasının bir kopyasıdır.[3]
Araştırmacıların ilgisini çekiyor çünkü daha iyi bilinen CRISPR/Cas9 sistemine benzer şekilde, DNA veya RNA'nın yüksek oranda hedeflenmiş modifikasyonlarını yapmak için kullanılabilir.[2] Cas12a, daha küçük ve daha basit olması, tracrRNA gerektirmemesi ve kesilen bölgede kör uçlar yerine yapışkan uçlar oluşturmasıyla Cas9'dan ayrılır. Bu ve diğer farklılıklar, onu belirli uygulamalarda daha uygun hale getirebilir. Temel araştırmalarda kullanımının ötesinde, CRISPR/Cas12a, genetik hastalıkların tedavisinde ve gen sürücülerinin uygulanmasında uygulamalara sahip olabilir.[2]
Yapı
Cas12a lokusu, karışık bir alfa/beta alanı, bir RuvC-I, ardından bir sarmal bölge, bir RuvC-II ve bir çinko parmak benzeri alan içerir.[4] Cas12a proteini, Cas9'un RuvC alanına benzer bir RuvC benzeri endonükleaz alanına sahiptir. Ayrıca, Cas12a bir HNH endonükleaz alanına sahip değildir ve Cas12a'nın N-terminali Cas9'un alfa sarmal tanıma lobuna sahip değildir.[1]
Cas12a CRISPR-Cas etki alanı mimarisi, Cas12a'nın işlevsel olarak benzersiz olduğunu ve Sınıf 2, tip V CRISPR sistemi olarak sınıflandırıldığını gösterir. Cas12a lokusları Cas1, Cas2 ve Cas4 proteinlerini tip II sistemlerden ziyade tip I ve III'e daha çok benzer şekilde kodlar. Veritabanı araştırmaları, birçok bakteri türünde Cas12a ailesi proteinlerinin bolluğunu göstermektedir.[1]
Fonksiyonel Cas12a tracrRNA'ya ihtiyaç duymaz, bu nedenle sadece crRNA gereklidir. Bu, genom düzenlemeye yarar çünkü Cas12a sadece Cas9'dan daha küçük değil, aynı zamanda daha küçük bir sgRNA molekülüne sahiptir (Cas9'un yaklaşık yarısı kadar nükleotid)
Cas12a-crRNA kompleksi, bir protospacer bitişik motifi (PAM) 5'-YTN-3'[7] tanımlayarak hedef DNA veya RNA'yı parçalar ("Y" bir pirimidindir[8] ve "N" herhangi bir nükleobazdır), Cas9 tarafından hedeflenen G açısından zengin PAM'nin aksine. PAM'nin tanımlanmasından sonra, Cas12a, 4 veya 5 nükleotid çıkıntısından oluşan, yapışkan uç benzeri bir DNA çift sarmallı kopması sunar.[4]
Mekanizma
CRISPR/Cas12a sistemi, bir Cas12a enziminden ve kompleksi bulan ve hedef DNA'yı parçalamak için çift sarmal üzerindeki doğru noktada konumlandıran bir kılavuz RNA'dan oluşur. CRISPR/Cas12a sistem etkinliğinin üç aşaması vardır:[5]
Adaptasyon: Cas1 ve Cas2 proteinleri, küçük DNA parçalarının CRISPR dizisine adaptasyonunu kolaylaştırır. .
crRNA'ların oluşumu: Cas proteinine rehberlik etmek için olgun crRNA'lar üreten pre-cr-RNA'ların işlenmesi.
Cas9 ve Cas12a
Cas9, DNA'yı kesmek için iki RNA molekülü gerektirirken Cas12a'nın bir taneye ihtiyacı vardır. Proteinler ayrıca DNA'yı farklı yerlerden keserek araştırmacılara bir düzenleme yeri seçerken daha fazla seçenek sunuyor. Cas9, bir DNA molekülündeki her iki zinciri de aynı pozisyonda keserek arkada kör uçlar bırakır. Cas12a, bir ipliği diğerinden daha uzun bırakarak yapışkan uçlar oluşturur. Yapışkan uçlar, homolog olmayan uç birleştirme veya DNA'nın homolog onarımı sırasında kör uçlardan farklı özelliklere sahiptir; bu, Cas9'a kıyasla gen yerleştirmeye çalışırken Cas12a'ya belirli avantajlar sağlar.[5] CRISPR/Cas9 sistemi genleri etkin bir şekilde devre dışı bırakabilse de, genleri eklemek veya bir knock-in oluşturmak zordur.[3] Cas12a tracrRNA'dan yoksundur, T açısından zengin bir PAM kullanır ve kademeli bir DNA DSB aracılığıyla DNA'yı parçalar.[6]
Referanslar
1-Makarova, Kira S., et al. "An updated evolutionary classification of CRISPR-Cas systems." Nature Reviews Microbiology (2015).
2."Even CRISPR". The Economist. ISSN 0013-0613. Retrieved 2016-05-03.
3."CRISPR-Based Genetic Engineering Gets a Kick in the Cas". Meta Science News. 2015-09-29. Archived from the original on 2017-10-22. Retrieved 2022-05-06.
4..Zetsche, Bernd; Gootenberg, Jonathan S.; Abudayyeh, Omar O.; Slaymaker, Ian M.; Makarova, Kira S.; Essletzbichler, Patrick; Volz, Sara E.; Joung, Julia; van der Oost, John; Regev, Aviv; Koonin, Eugene V.; Zhang, Feng (October 2015). "Cpf1 Is a Single RNA-Guided Endonuclease of a Class 2 CRISPR-Cas System". Cell. 163 (3): 759–771. doi:10.1016/j.cell.2015.09.038.
5.Ledford, Heidi (2015). "Alternative CRISPR system could improve genome editing". Nature. 526 (7571): 17. doi:10.1038/nature.2015.18432. PMID 26432219.
6. "Cpf1 Moves in on Cas9 for Next-Gen CRISPR Genome Editing". epigenie.com. Retrieved 2022-05-06.
7. Fonfara I, Richter H, Bratovič M, Le Rhun A, Charpentier E (2016). "The CRISPR-associated DNA-cleaving enzyme Cas12a also processes precursor CRISPR RNA". Nature. 532 (7600): 517–521. doi:10.1038/nature17945.
8."Nucleotide Codes, Amino Acid Codes, and Genetic Codes". KEGG: Kyoto Encyclopedia of Genes and Genomes. July 15, 2014. Retrieved 2022-05-06.


Yorumlar
Yorum Gönder